【連載】基礎から応用までよくわかる組織透明化技術 「第2回 高解像・高輝度蛍光イメージングのための透明化法、SeeDB2」
本記事は、和光純薬時報 Vol.87 No.3(2019年7月号)において、九州大学大学院医学研究院 今井 猛様に執筆いただいたものです。

はじめに
近年、様々なレーザー顕微鏡が開発されているが、生体組織を3次元的に捉えることは容易ではない。生体組織は光の散乱や吸収が生じ、深部に行けば行くほど観察が難しくなるためである。近年、こうした問題を克服する手段として、固定した生体組織を透明にする手法が注目されている1, 2)。いずれも、光の吸収および散乱を減らすことで深部の観察を実現している。今や、組織や臓器の全体像を3次元的にとらえることも容易になってきた。
このように、透明化というと、多くの読者は大きな組織や臓器の観察に使う手法と捉えているかもしれない。それはそれで間違っていないのだが、透明化法は、「高解像度」イメージングにおいても非常に有用なツールである。そこで本稿では、透明化によって「高解像度」の3次元蛍光画像を得るための基礎知識と、我々が開発したSeeDB2について解説したい。
光学顕微鏡の分解能と開口数(NA)、球面収差
はじめに、高解像イメージングについて考える上では、顕微鏡の分解能について説明しなければならない。共焦点顕微鏡などのレーザー蛍光顕微鏡において、水平方向の分解能dは以下のレーリーの式によって表される。
d = 0.61λ/NA
ここで、λは光の波長、NAは対物レンズの開口数(Numerical Aperture)を示す。この式によれば、dの値を小さくするにはNAの値を大きくすれば良いことがわかる。蛍光イメージングにおいて分解能を決めるのは倍率ではなく、NAである。NAの値は
NA = n sinα
で定義される。ここで、n は浸液の屈折率、αは開口角の1/2の値である。このため、高開口数を持つ対物レンズの多くは、ドライレンズではなく、高屈折率の浸液、すなわちオイル(屈折率 1.52)やグリセリン(屈折率 1.45-1.46)を用いるように設計されている。これにより、200 nm程度の分解能が実現できる。NAが大きいと良いのは分解能だけではない。NAが大きいほど対物レンズに入ってくる光の量が多くなるため、明るい像が得られる。明るさはNAの二乗に比例するため、例えばNA1.4の油浸レンズはNA1.0の水浸レンズの2倍程度明るい。
このように、細かい構造を明るく観察するにはNAの大きな対物レンズが有効である。一方でうまく使用しないと像がボケて暗くなりやすいという問題がある。油浸レンズを用いて「標本の表面」を観察する場合、焦点位置までには屈折率1.52の浸液と、同じく屈折率1.52のカバーガラスがあるため、光はまっすぐ進んで、蛍光シグナルもまっすぐに戻ってくることになる。
しかしながら、少し深いところを観察しようとすると大きな問題が生じる。標本の屈折率は、水の1.33から、グリセリン系封入剤の1.46の間であることが多い(図1A)。この場合、カバーガラスと標本の界面では屈折率の違いから屈折が生じ、本来1点に収束するはずであった励起光は広がってしまうことになる。この結果、像がボケる。球面収差によるボケは水平方向(x-y)よりも軸方向(z)でより顕著である。
また、蛍光シグナルが対物レンズに戻ってくる際にも屈折し、ピンホールを通って検出器に戻ってくる光量が減ってしまう。この結果、像が暗くなる。このように、標本の屈折率の不一致から生じる収差「球面収差」があると、像がボケ、暗くなる。標本深部ではこの球面収差が大きくなるため、従来、高NAレンズを使って標本深部を観察することは困難であった。
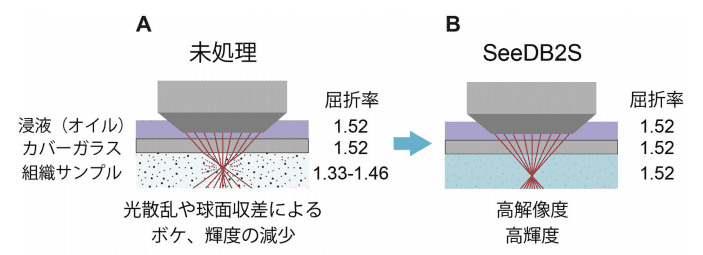
図1.SeeDB2を用いた高解像イメージングの原理3)
高解像度が要求される実験では、グリセリン浸(NA1.3以上)や油浸(NA1.4以上)レンズが有効である。しかしながら、屈折率が最適化されていない従来の封入剤では標本深部において球面収差が生じ、設計通りの分解能を得ることができない(A)。SeeDB2を用いて標本の屈折率を浸液と一致させることで、球面収差を抑え、高解像度・高輝度の画像を得ることができる(B)。SeeDB2による高解像・高輝度イメージング
球面収差を最小限にし、高NAレンズでの深部観察を実現するため、我々は新しい透明化試薬SeeDB2を開発した3)。SeeDB2は油浸レンズ(SeeDB2Sを使用、屈折率1.52)もしくはグリセリン浸レンズ(SeeDB2Gを使用、屈折率1.45-1.46)の浸液と屈折率を一致させることができるため、球面収差を極限まで減らすことができる(図1B)。
これにより、深部でも分解能を一定に保つことができるのみならず(図2A)、輝度も深さによらずほぼ一定となる(図2B)。また従来の透明化液のように標本の伸縮をもたらすことがないため、微細形態が高度に保持されている。
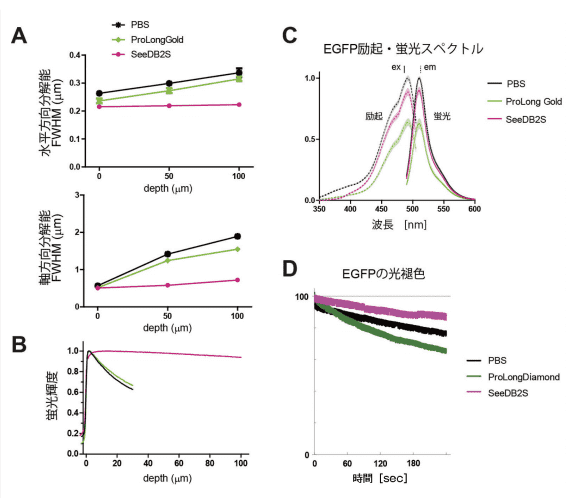
図2.SeeDB2による高解像・高輝度イメージング(A-Cは文献3)より転載)
(A) 水平方向(x-y)および軸方向(z)の分解能(FWHM)。SeeDB2を用いると標本深部でも分解能が一定である。
(B) 球面収差を減らすと、深部においても高輝度の観察が可能となる。図はローダミン溶液を共焦点顕微鏡で観察し(油浸レンズ使用)、輝度を測定したもの。SeeDB2Sを使うと深部 までほぼ一定の輝度が得られる。
(C) SeeDB2は蛍光タンパク質の蛍光輝度がほとんど消光しない。一般的な市販の封入剤と比較。
(D) SeeDB2では蛍光タンパク質の光褪色も抑えられている。蛍光タンパク質用に市販されている封入剤と比較。
こうしたことから、SeeDB2は微細形態の高解像観察には最適である。SeeDB2は大きな臓器・組織の観察だけでなく、比較的小さな昆虫などの組織や培養細胞の高解像観察においても大きな威力を発揮する。SeeDB2のプロトコルは我々のウェブサイトSeeDB Resources で公開されているほか4)、bio-protocol誌にも詳細なプロトコルを発表している5)。調製済みのSeeDB2試薬は市販されている。SeeDB2S(屈折率1.52)を観察時の封入剤として使用する場合には、Super Clear Mountという名称で市販されているものをそのまま使用すればいい。
分解能だけでなく、蛍光タンパク質の安定性もSeeDB2S / Super Clear Mountの特筆すべき利点である。意外と知られていない点であるが、市販の封入剤はフルオレセインやローダミン、Alexa色素といった合成色素の安定性という観点で最適化されているため、蛍光タンパク質の安定性は必ずしも良くない。実際、汎用されているグリセリン系のマウント剤で蛍光タンパク質標本をマウントすると、EGFPやEYFPの蛍光が3-7割も減少してしまう3)。更に、従来市販されている封入剤では蛍光タンパク質の光褪色も大きい。
これに対し、SeeDB2S / Super Clear Mountを用いると、いずれの蛍光タンパク質もほとんど蛍光輝度が変化せず、光褪色も極めて起こりにくい(図2C, D)。こうしたことから、蛍光タンパク質の観察には最適である。一方、市販のマウント剤と比べると、DAPIやAlexa色素の光褪色が生じやすいのは欠点と言える。
共焦点顕微鏡、超解像顕微鏡への応用
SeeDB2が極めて有効な応用例の1つとして、神経細胞のシナプスの定量解析が挙げられる。多くの神経細胞において、興奮性のシナプス入力は樹状突起スパインと呼ばれる小突起に限局している。樹状突起スパインはシナプスの可塑性に伴って大きさが変化するほか、その密度や形態の異常が精神疾患につながることも示唆されている。
こうしたことから、2光子励起顕微鏡や共焦点顕微鏡を用いた樹状突起スパインの定量解析が広く行われている。しかしながら、樹状突起スパインは頭部の直径が0.1-1 µm程度と極めて小さく、正確な定量は容易ではない。われわれが開発したSeeDB2Sを用いると、共焦点顕微鏡と油浸レンズを使って標本深部まで高解像画像を得ることができるようになった。高NA対物レンズは作動距離が100-300 µm程度であり、その範囲内であれば高解像度の画像を得ることができる(図3)。
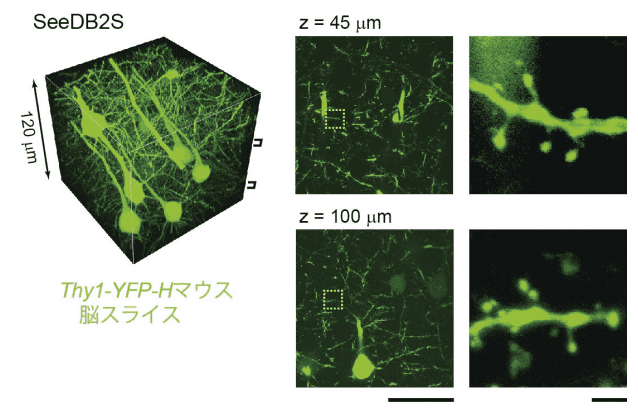
図3.共焦点顕微鏡による深部高解像観察(文献3)より転載)
Thy1-YFP H line マウス脳スライスをSeeDB2Sで処理し、NA1.4の油浸レンズを使用して共焦点顕微鏡観察を行った。スライスの浅い部分と深い部分でほぼ一定の分解能が得られる。またレーザーパワーを変えなくてもほぼ一定の輝度が得られる。右に示すのは樹状突起スパイン。スケールバーは左が50 µm、右が2 µm。
共焦点顕微鏡だけでなく、超解像顕微鏡でも深部の観察が可能である。STED顕微鏡(分解能約50 nm)やAiryscan(分解能 120-150 nm、図4)でも深さ100 µm超まで高解像観察が可能である。神経突起の解析を行う上では、x-y方向の分解能だけでなく、z軸方向の分解能も極めて重要である。SeeDB2を用いると、従来法に比べてz軸方向の分解能が劇的に改善するため、神経突起のトレーシングや樹状突起スパインの定量解析に有効である。
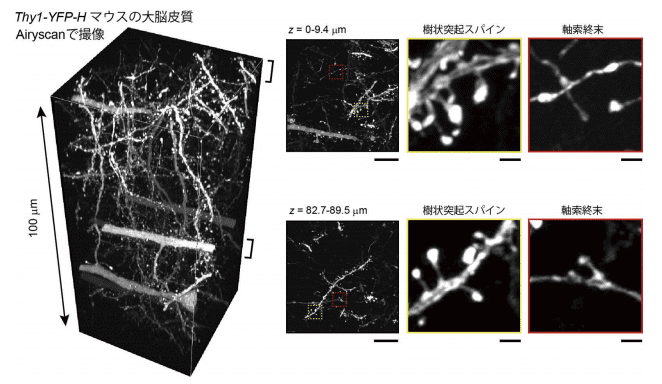
図4.Airyscan (Zeiss社)による脳スライスの観察例(文献3)より転載)
スケールバーは左が10 µm、中央と右が1 µm。動画をYouTubeにて公開(https://youtu.be/TakrFLY7pU4)。
SeeDB2S / Super Clear Mount は、厚い標本だけでなく、培養細胞等の超解像イメージングのための封入剤としても極めて有効である。図5はHEK293細胞をSR-SIMで撮像したものであるが、球面収差を減らすことで膜やオルガネラの構造がより鮮明に観察できている。
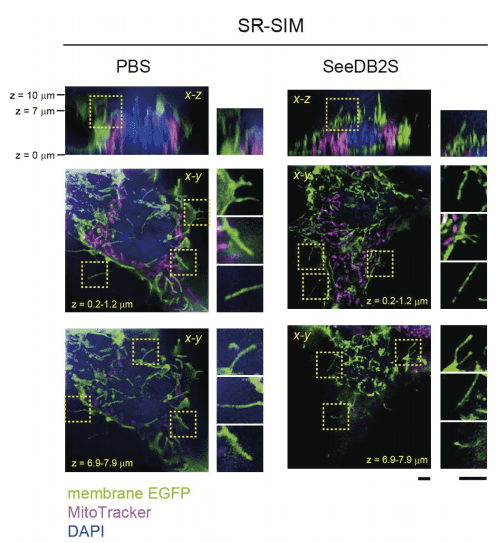
図5.SR-SIM(Zeiss社)による培養細胞(HEK293細胞)の超解像イメージング例(文献3) より転載)
スケールバーは2 µm。
Tetbowを用いた神経細胞の多色標識
蛍光イメージングにおいて、透明化試薬によるサンプル調製も重要であるが、同じくらい重要なのは標本の標識である。見たい対象物が高輝度染色されていなければ、いくらサンプル調製を工夫しても鮮明な画像を得ることはできない。ここでは、われわれが神経回路解析のために新たに開発した手法、Tetbowについて紹介したい。
脳内では多くの神経突起が絡まり合っており、その配線の全容を明らかにすることは容易ではない。蛍光タンパク質を使って一部の神経細胞を標識すれば、個々の突起の配線の様子を観察できるが、多くの神経細胞が同じ色で染色されてしまうと、それぞれを区別するのが困難になってしまう。
こうした問題を克服するため、3種類の蛍光タンパク質を組み合わせ、神経細胞を数10種類もの異なる色で染め分ける方法、Brainbowが報告されている6, 7)。しかしながら、Brainbowは蛍光輝度が十分ではなかったため、実用的ではなかった。そこで、われわれはTet-Offシステムを使うなどの工夫をし、従来よりも一桁程度高輝度な多色標識法Tetbow法を開発した(図6)8)。
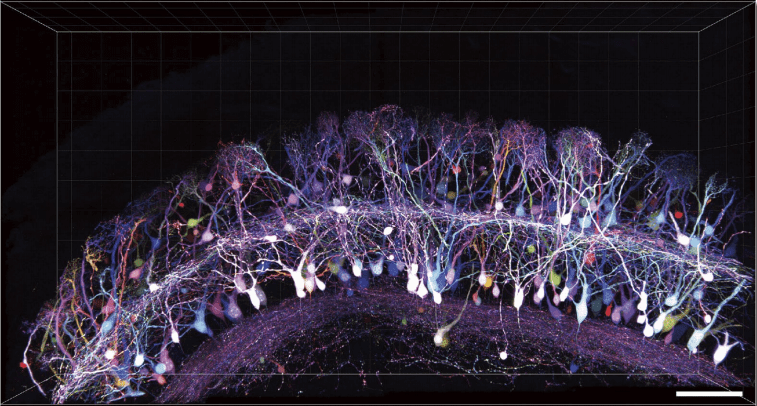
図6.Tetbowによる神経細胞の多色蛍光イメージング(文献5) より転載)
プラスミドを用いたin utero electroporation法によって、嗅球僧房細胞をTetbowで標識した。スケールバーは100 µm。動画をYouTubeにて公開(https://youtu.be/EHato3VVQTs)。Tetbow法はプラスミドやウイルスベクターを用いて神経細胞に導入可能である。CFP、YFP、RFPなどの蛍光タンパク質はいずれもSeeDB2中で極めて安定であるため、SeeDB2を用いた透明化が最適である。また、蛍光タンパク質の代わりにSNAP、HaloTag、CLIPといったケミカルタグを使えば、任意の合成色素を使って多色標識を実現できる。実用に耐える輝度を実現できたことで、多色標識は今後の神経回路解析のための有用な手段になるものと期待される。
おわりに
本稿では、特にSeeDB2にフォーカスして、高解像・高輝度イメージングを行うための基礎知識と注意点について解説した。対物レンズのNAや標本の屈折率に留意して標本を調製することで、貴重な標本からより多くの情報を正確に引き出すことができる。本稿が今後の蛍光イメージング実験の一助となれば幸いである。
参考文献
- Richardson, D. S. and Lichtman, J. W. : Cell, 162(2), 246 (2015). DOI: 10.1016/j.cell.2015.06.067
- Richardson, D. S. and Lichtman, J. W. : Cell, 171(2), 496.e1. (2017). DOI: 10.1016/j.cell.2017.09.025
- Ke, M. T. et al. : Cell Rep. 14, 2718 (2016). DOI: 10.1016/j.celrep.2016.02.057
- SeeDB Resources, https://sites.google.com/site/seedbresources/
- Ke, M. T. et al. : Bio-protocol, 8(20), e3046 (2018). DOI: 10.21769/BioProtoc.3046
- Livet, J. et al. : Nature, 450, 56 (2007). DOI: 10.1038/nature06293
- Cai, D. et al. : Nat. Meth., 10, 540 (2013). DOI: 10.1038/nmeth.2450
- Sakaguchi, R. et al . : eLife, 7, e40350 (2018). DOI:10.7554/eLife.40350









