クロマチン免疫沈降法(ChIP) - 原理/方法をわかりやすく解説
クロマチン免疫沈降法(ChIP)とは?
クロマチン免疫沈降法(Chromatin Immunoprecipitation:ChIP)は、DNA-タンパク質の相互作用の解析に用いられます。遺伝子発現調節やクロマチン構造変化など、エピジェネティクス研究を進める上で極めて重要です。特定のタンパク質とDNAの相互作用の解析だけでなく、複数のタンパク質とDNAの相互作用の解析、タンパク質のゲノム全体における相互作用の解析など、汎用性の高い方法です。
ChIPプロトコールの主なステップは以下の通りです。
- DNAとタンパク質の固定(クロスリンク)
- 細胞溶解
- DNAの断片化(ソニケーション法/酵素法)
- 抗体を用いた免疫沈降
- 脱クロスリンク
- DNA精製
- 濃縮したDNAの解析(qPCR、NGS、マイクロアレイ)
本記事では、クロマチン免疫沈降法の原理、方法、実験のヒントやコツをわかりやすく解説します。
原理
はじめに目的の細胞を回収し、ホルムアルデヒドを用いてDNAとタンパク質を架橋します。次に、超音波処理や制限酵素処理によりDNA-タンパク質の複合体を断片化します。これを標的タンパク質に対する抗体で免疫沈降を行い、DNA断片の精製・回収します。得られたDNAを鋳型にして、目的のDNA配列特異的プライマーを用いたPCR、もしくは定量的PCR(qPCR)で解析を行います。これにより、ヒストンや転写因子といったタンパク質のクロマチン領域における結合量を調べることができます。
また、ChIPとマイクロアレイを組み合わせたChIP-on-chipや、ChIPと次世代シーケンサーを組み合わせたChIP-seq(chromatin immunoprecipitation with high-throughput sequencing)も多く用いられており、特定の転写因子の結合領域やヒストン修飾の状態をゲノムワイドに解析できます。
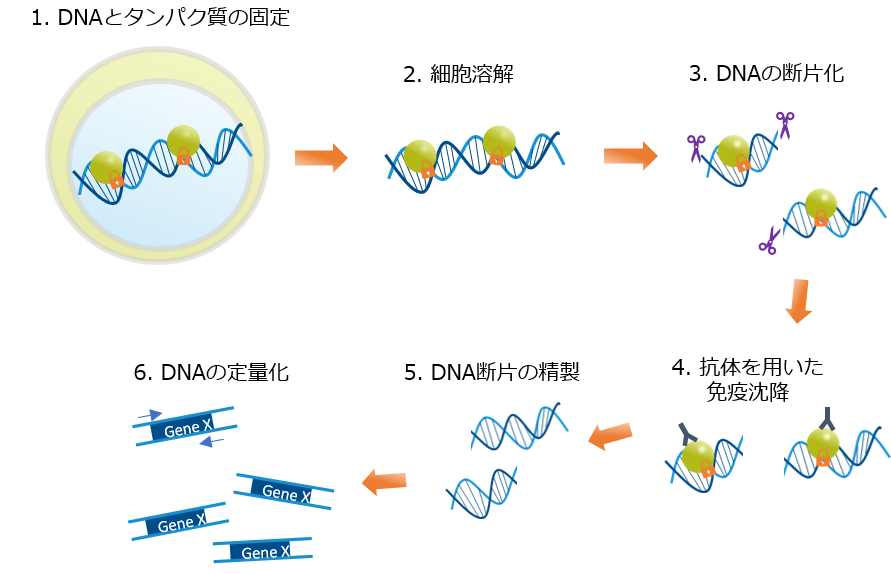
手順

1. DNAとタンパク質の固定(クロスリンク)
DNAとタンパク質の固定には、一般的にホルムアルデヒドが架橋剤として使用され、グリシンの添加により反応を停止させます。架橋剤は細胞内に浸透し、DNAとタンパク質の相互作用を安定化させることで解析が可能になります。ただし、架橋剤によるDNAとタンパク質の相互作用は、可逆性でなければいけません。
【実験のヒントとコツ】
・架橋が不十分あるいは過剰な場合、バックグラウンドを上昇させる要因となり、DNAのロスにつながります。架橋が不十分な場合、DNAとタンパク質が脱会合し、免疫沈降による回収量が低下します。架橋が過剰な場合、エピトープがマスクされることで細胞溶解の効率が低下し、超音波処理の妨げになるとともに、脱クロスリンクができない可能性があります。
・クロスリンクは、架橋剤の濃度と架橋時間の両方が重要です。
・架橋剤の種類は、解析する相互作用により異なります。
【推奨製品】
2. 細胞溶解
溶解液により膜透過処理を行い、細胞成分を分離することで、細胞からDNAとタンパク質の複合体を抽出します。サイトゾルに含まれるタンパク質を除去することにより、バックグラウンドが低減され、感度が上がります。
【実験のヒントとコツ】
- 透過処理の前後で、細胞溶解の効率を顕微鏡で確認してください(溶解の効率は細胞の種類により異なります)。
- サンプルは常に氷上に置いてください。
【推奨製品】

3. DNAの断片化(ソニケーション法/酵素法)
DNAの断片化は、コントロールが難しいステップの1つであり、ChIPの解像度に大きく影響します。超音波処理(ソニケーション)または酵素処理により、DNAを断片化します。理想的な断片のサイズは100~500 bpです。
超音波破砕と酵素消化には、それぞれメリットとデメリットがあります。細胞株に応じて最適化する必要があります。
【実験のヒントとコツ】
- 過剰なせん断は、エピトープにダメージを与える可能性があります。また、断片が小さくなることにより、qPCRにおけるプライマーの結合を低下させるリスクがあります。
- せん断の不足は、免疫沈降における非特異的な結合を増大させます。
- DNAの断片化を初めて行う際は、条件検討を行い、最適化してください。
- せん断後のDNA断片の長さが、100~500 bpに収まっていることを確認してください。
- せん断効率を安定化させるために、常に氷上で操作を行い、また、サンプルの泡立ちを避けてください。
- inputサンプル(定量分析用のコントロール)を用意してください。定量分析する際に、inputサンプルのDNA量に対する免疫沈降したサンプルのDNA量を比較します。
【推奨製品】
| 超音波破砕 | 酵素消化 | |
|---|---|---|
| メリット |
|
|
| デメリット |
|
|
4. 抗体を用いた免疫沈降
断片化したDNA-タンパク質の複合体を抗体により免疫沈降し、その他の細胞物質を排除します。 標的タンパク質を認識する抗体を、断片化したDNA-タンパク質複合体溶液に加え、抗原-抗体の複合体を形成させます。免疫沈降(IP)時間は4℃,オーバーナイトで行うことが多いですが、1時間程度の短時間で行うことができる場合もあります。その後、ビーズを用いて抗原-抗体の複合体を回収し、洗浄を行います。
【実験のヒントとコツ】
- 抗体の選択が実験の成功に重要なポイントです。標的タンパク質を認識する検証済の抗体を用いることをおすすめします。抗体の選択については、下記の「実験を成功させるポイント」で詳しく解説しています。
【推奨製品】
5. 脱クロスリンク
免疫沈降により回収されたDNA-タンパク質複合体は、脱架橋し、DNAを単離する必要があります。一般的に、高温インキュベーションもしくはタンパク質消化により脱架橋を行います。ホルムアルデヒドを用いて架橋を行った場合は、SDSの存在下で高温インキュベーションを行うことで、加水分解されます。
【推奨製品】
6. DNA精製
単離されたDNAは、RNase処理や、フェノールクロロホルム処理、DNA精製用のスピンカラムキットを用いて精製します。RNase処理は、より純粋なDNAを得るために用いられます。フェノールクロロホルム処理、DNA精製用のスピンカラムキットは、残留タンパク質を除去するために用いられます。
【推奨製品】
7. 濃縮したDNAの解析(qPCR、NGS、マイクロアレイ)
精製したDNAを定量します。
qPCRでは、目的タンパク質が特定のDNA領域に局在するかどうかを調べることができます。qPCR試薬としては、SYBR Green蛍光色素が最も広く使用されています。
SYBR Greenは、二本鎖DNA(dsDNA)に結合することで蛍光を発する試薬(インターカレーター)です。インターカレーターは、PCRで合成した二本鎖DNAに結合し、励起光を照射することにより、蛍光を発します。この蛍光強度を検出することにより、PCR産物の生成量をモニターできます。
qPCR結果から定量値を計算する方法は、以下の通りです。
インプット(Input)に含まれるDNA量に対する、免疫沈降(IP)後のDNAの回収率(IP%)を算出します。
IP%=2(InputのCt値−IPのCt値) ×100×濃縮率
Input:定量分析用のコントロールサンプル。DNA断片化後に免疫沈降を行わないサンプルとして回収する。IP:免疫沈降の手順を行ったサンプル。
Ct値:qPCR反応の閾値と、増幅による蛍光シグナルが交差する時点のサイクル数
濃縮率:Inputサンプルに対するIPサンプルの濃縮率
【実験のヒントとコツ】
- ターゲットとなるDNA配列に合わせてプライマーを設計し、90~105%の効率であることを確認してください。
- ChIPシーケンスを行う場合においても、事前にqPCRを行い、実験が機能していることを確認してください。
【推奨製品】
- GeneAce qPCR Mixシリーズ
- 1つのエピトープに反応
- 特異性が高い
- 複数のエピトープに反応
- 特異性が低い
- 非特異的な結合が少ない
- 視認性が良好
- 再現性が高い
- 表面積が広く、結合能が高い(多孔性)
- 必要な装置が少ない
- 低コスト
- コストが高い
- 非多孔のため結合能が低い
- 磁気ラックが必要
- 事前精製、ブロッキングが必要
- 視認性が低い
- サンプルロスの確率が高い
ChIPを成功させるポイント
抗体の選択
ChIP実験を成功させるためには、適切な抗体選択が極めて重要です。ChIPで使用する抗体は、DNA-タンパク質複合体のエピトープを認識できる必要があります。
ChIPに最適な抗体は、ChIP検証済みの抗体です。ChIP検証済みがない場合は、IP検証済み抗体を選択します。しかし、IP検証済済み抗体が必ずしもChIPで機能するとは限りません。また、ウエスタンブロット(WB)、免疫蛍光染色(IF)などに使用できる抗体でもChIPに使用できない場合があります。
そのため、抗体の濃度やIPの洗浄条件などを検討した上で決定する必要があります。
抗体を選択する際には、モノクローナル抗体とポリクローナル抗体の理解が重要です。
ChIP実験には、モノクローナル抗体とポリクローナル抗体のどちらも使用できます。
それぞれの特長を以下に示します。
| モノクローナル抗体 | ポリクローナル抗体 | |
|---|---|---|
| 由来 | ハイブリドーマ | 抗血清 |
| 特性 | ||
| 再現性 | ロット間の再現性が高い | ロット間で性能が異なることがある |
モノクローナル抗体は、エピトープが遮断されている、あるいはDNA-タンパク質のクロスリンクの過程で構造変化が生じた場合、標的タンパク質を認識することができません。一方、ポリクローナル抗体は、複数のエピトープに反応するため、エピトープが遮断された場合に有効な場合があります。しかし、ポリクローナル抗体は、複数のエピトープに反応することから、非特異的な結合が起こる可能性が高くなります。
モノクローナル抗体、ポリクローナル抗体のいずれを用いる場合においても、特異性を示すデータや様々なアプリケーションにおけるデータなど、十分に検証されている抗体を用いることが理想的です。
当社では、ChIPに使用するモノクローナル抗体を幅広く取り扱っています。
抗体ラインアップはこちら
ビーズの選択
ChIP実験では、抗体をどのビーズに結合させるか検討する必要があります。
選択肢としては、抗体を直接磁気ビーズもしくはアガロースビーズに結合させる、または、Protein Aや Protein G、Protein A/Gビーズに固定する方法があります。
本来のChIP手法ではアガロースビーズを用いられますが、現在では磁気ビーズを使う研究者が増加している傾向にあります。
磁気ビーズとアガロースビーズのメリット・デメリットを以下に示します。
| 磁気ビーズ | アガロースビーズ | |
|---|---|---|
| メリット | ||
| デメリット |
細胞数
ChIPに必要な細胞の数は、標的タンパク質の存在量によって変わります。
細胞数の目安は以下の通りです。
当社では、細胞培養に最適な細胞培地を各種ラインアップしています。
| 標的タンパク質の存在量 | 例 | 細胞数 |
|---|---|---|
| 多 | 修飾ヒストン、RNA PoⅡ | 104 |
| 中 | 基本転写因子、修飾因子 | 105~106 |
| 低 | 配列特異的転写因子 | 106~107 |
| 低 | アクセサリー因子 | <107 |
ChIPによるエピジェネティクス研究の進展

エピジェネティクス研究は、クロマチンの構造変化を引き起こすDNAのメチル化やヒストン修飾を対象としており、DNA-タンパク質の相互作用に焦点が当てられています。クロマチン免疫沈降法(ChIP)は、これらエピジェネティックな遺伝子発現パターンや制御機構の解析に極めて重要です。
ChIPは、qPCR解析だけでなく、マイクロアレイ分析(ChIP-on-chip)、シーケンス解析(ChIP-seq)といったゲノムワイドな解析が可能です。しかし、ChIPに必要な細胞数を回収することが実験のハードルとなります。
近年、ATAC-seq(Assay for Transposase-Accessible Chromatin with high throughput sequencing)の開発により、500~5000個の細胞数で、全遺伝子領域における転写活性化部位とその頻度の解析が可能になりました。また、クロマチンから直接NGSライブラリの構築が可能なChIPmentationという技術も開発されました。少ない細胞数にもかかわらず、短時間で高品質なデータを得ることが可能なため、がんなどの希少な細胞を用いたエピジェネティクス研究に用いられます。
このように、エピジェネティックな遺伝子発現パターンや制御機構を研究するための技術が開発されており、さらなるエピジェネティクス研究の進展が期待されています。




