【テクニカルレポート】高性能 Bisulfite の開発
本記事は、和光純薬時報 Vol.84 No.4(2016年10月号)において和光純薬工業 ライフサイエンス研究所 林田 幸信が執筆したものです。
生体内におけるゲノムDNA のメチル化は遺伝子発現の制御に関わり、メチル化のパターンの差異が、発生、分化、がん等の疾患に関係することが報告されています。ゲノムDNA のメチル化解析は病気の原因解明や予防、医薬品の開発、再生医療の研究等において重要な役割を持っています。
現在、DNA 塩基配列中のメチル化シトシンを測定する方法としては Bisulfite 法が一般的な方法として普及しています。
従来の一般的なBisulfite 法はDNAの分解が激しいため、PCR 増幅鎖長の設定を短くする必要があります。また PCR 増幅する遺伝子領域によっては非メチル化シトシンからウラシルへの変換率が高くないため、メチル化シトシンの検出精度が低い等の問題を有していました。そのため、遺伝子配列の影響を低減した非メチル化シトシンのウラシルへの変換効率が高い Bisulfite 法の開発が望まれていました。
図1 はLambda DNA を鋳型として EpiSight™ Bisulfite Conversion Kit Ver.2 により Bisulfite 反応後、910bp という Bisulfite PCR では長い鎖長を増幅し、塩基配列を解読した結果です。Lambda DNA はすべてのシトシンは非メチル化であり、Bisulfite 反応後はすべてウラシルに変換され、PCR 増幅産物はシトシンが存在しないと予想されます。
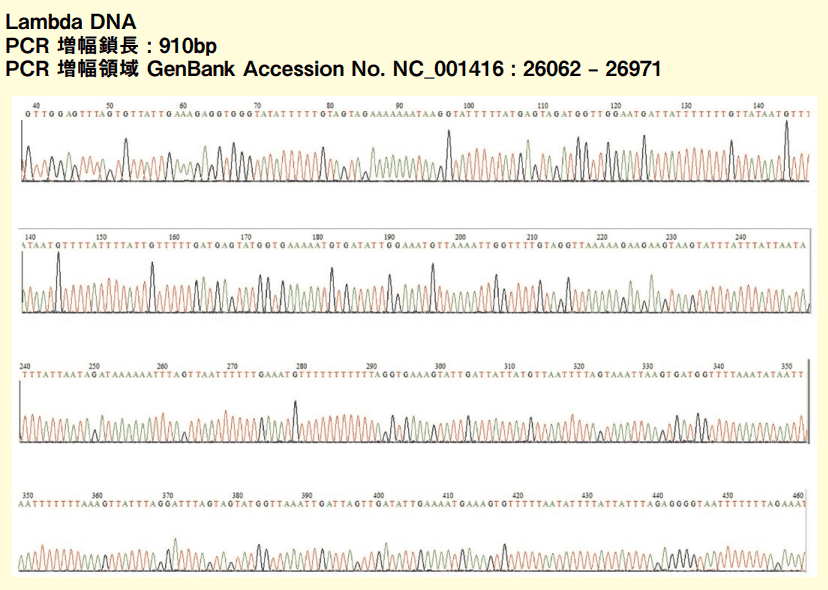
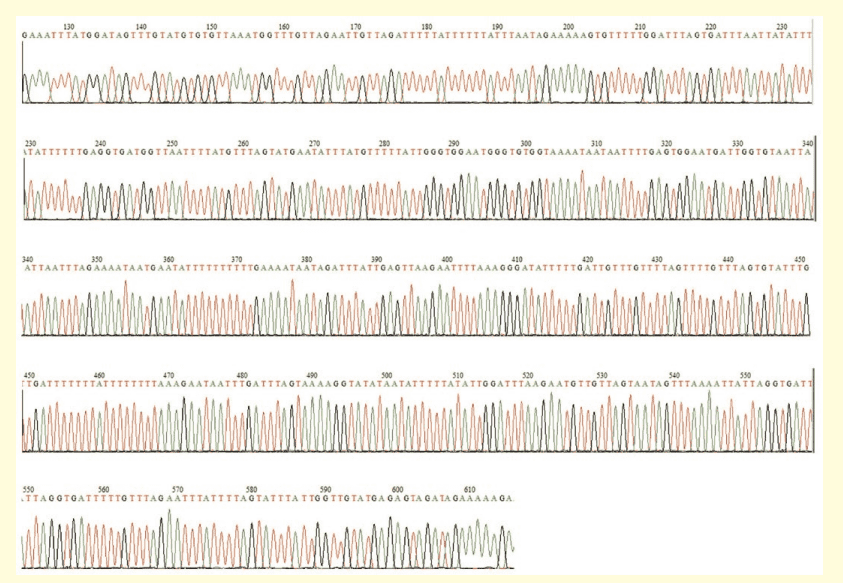
図1.Lambda DNA PCR 増幅産物塩基配列解読結果
図1 の波形チャートにはシトシンは存在せず、150 箇所あるシトシンはすべてチミンに変換されています。EpiSight™ Bisulfite Conversion Kit Ver.2 は Enhancer を Bisulfite 反応時に添加することにより一般的な Bisulfite 法ではDNAの分解が激しくて条件設定できなかった 900bp 以上の PCR 産物の増幅に成功しており、また非メチル化シトシンのウラシルへの変換率もほぼ100%に近いデータが得られています。
今回、マウスES 細胞(胚性幹細胞)及び M1 細胞(白血病由来細胞)ゲノムDNA を鋳型としてEpiSight™ Bisulfite Conversion Kit Ver.2 により Bisulfite 反応後、幹細胞未分化マーカーである Fgf4 プロモーター領域の PCR 増幅を試みました。PCR 増幅鎖長は 521bp、CpG ジヌクレオチド数は 61 箇所という Bisulfite PCR としては長い増幅鎖長であり、GC 含量も高いことから立体構造の影響も大きいと予想される領域を選択しました。
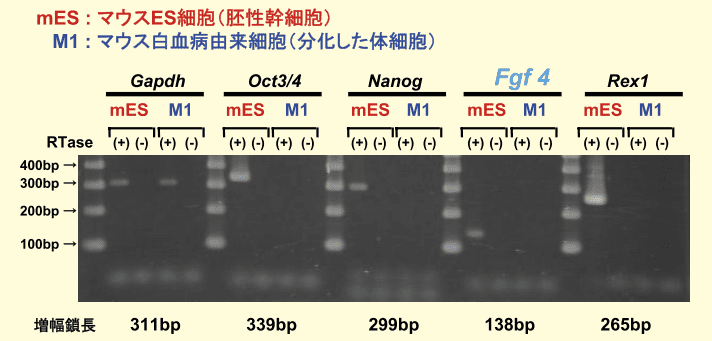
図2.幹細胞未分化マーカー発現量解析
ES 細胞及び M1 細胞の幹細胞未分化マーカーの発現解析を図 2 に示しています。ES 細胞は幹細胞ですので幹細胞未分化マーカーである Fgf4 の mRNAは発現しており、プロモーター領域は非メチル化状態、一方 M1 細胞は分化した体細胞ですので Fgf4 の mRNA は発現しておらず、プロモーター領域はメチル化されていると予想されます。ES 細胞及び M1 細胞の増幅産物の塩基配列を解読した結果、ES 細胞では CpG ジヌクレオチドも含めてほぼすべてのシトシンはウラシル(チミン)に変換されていました(図 3 − 1)。一方、M1 細胞では CpG ジヌクレオチド以外のシトシンはウラシル(チミン)に変換されており、CpG ジヌクレオチドのシトシンはシトシンでした(図 3 − 2)。
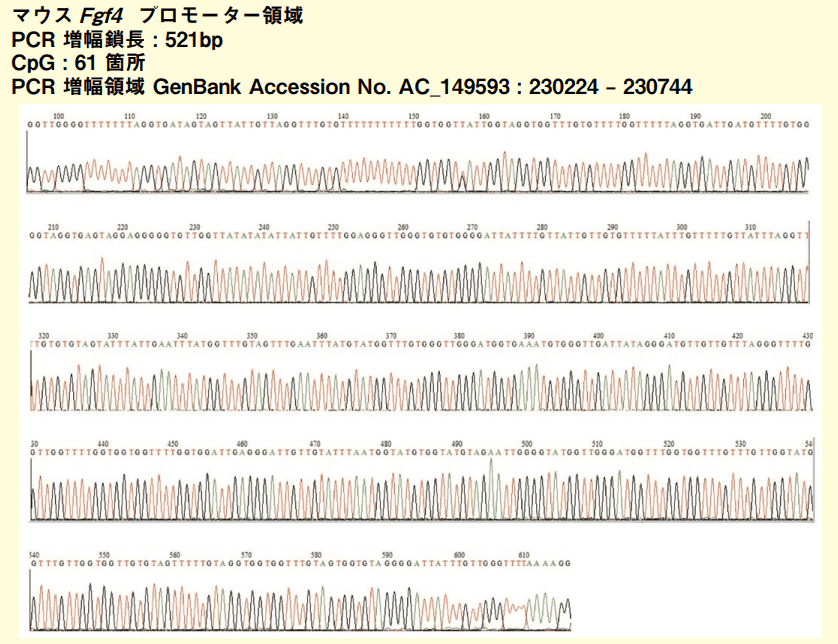
図3−1.マウス ES 細胞 PCR 増幅産物塩基配列解読結果
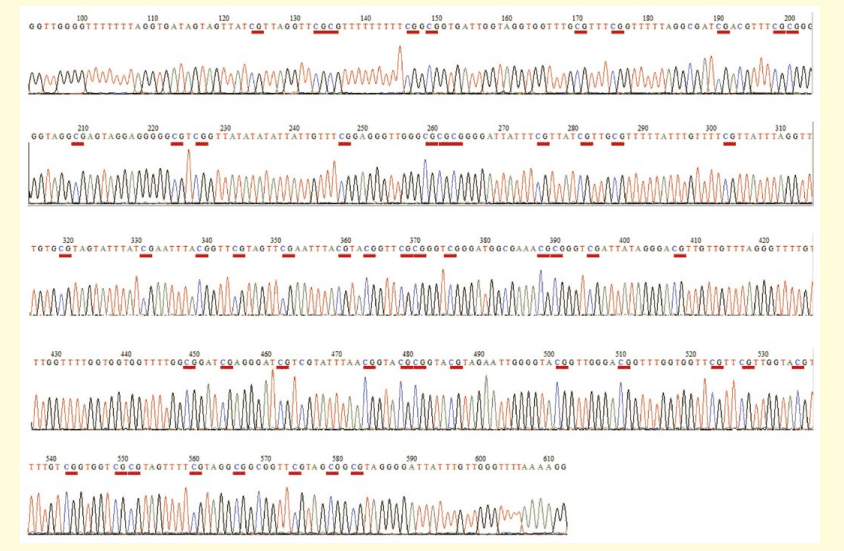
図3−2.マウス M1 細胞 PCR 増幅産物塩基配列解読結果
図4 は ES 細胞及び M1 細胞の PCR 増幅産物をクローニング後、各 11 サンプル塩基配列を解読した結果です。今回の結果は Fgf4 mRNA の発現量を反映しています。

図4.マウス ES 及び M1 細胞Fgf4 プロモーター領域メチル化解析結果
EpiSight™ Bisulfite Conversion Kit Ver.2 はスピンカラムの使用により精製過程の簡便化及び短時間化を実現しておりさらに使いやすくなっています。
ぜひとも検討していただければと思います。




