【総説】Phos-tag 技術を用いたタンパク質のリン酸化解析
本記事は、和光純薬時報 Vol.87 No.2(2019年6月号)において、広島大学大学院医歯薬保健学研究科 医薬分子機能科学研究室 木下 恵美子様、木下 英司様、小池 透様に執筆いただいたものです。
はじめに
細胞内で生命活動を担う多くのタンパク質は、キナーゼによるリン酸化とフォスファターゼによる脱リン酸化を繰り返し、活性のONとOFFを切り替えている。しかし、キナーゼやフォスファターゼの異常によりこの活性制御のバランスが崩れると、様々な病気が引き起こされることが知られている。特にがんでは種々のキナーゼの異常が報告されている。それゆえに、タンパク質のリン酸化研究は、様々な疾患の原因究明やキナーゼ阻害剤(分子標的薬)の開発につながる非常に重要な研究分野となっている。
筆者らの研究グループは、リン酸基(-PO32-)を補足する機能性分子Phos-tag(フォスタグ)を用いた、タンパク質のリン酸化解析技術の開発を行っている。本稿では、これまでに開発したPhos-tagを基盤とした技術のうち、多くの生命科学研究者に利用されるようになった3つについて紹介したい。
Phos-tag SDS-PAGEによるリン酸化タンパク質の解析法
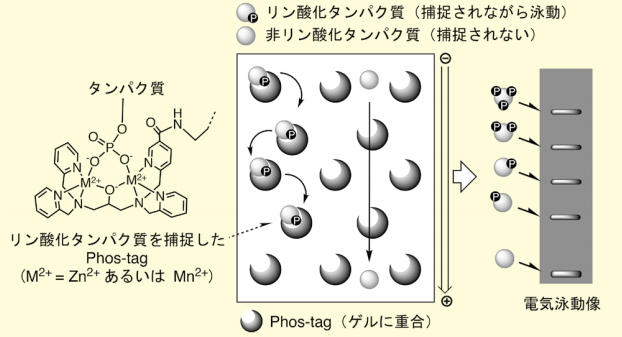
図1.Phos-tag SDS-PAGEの概略
1つ目は、SDS-PAGEを用いてリン酸化タンパク質と非リン酸化タンパク質を分離するリン酸基アフィニティ電気泳動法、Phos-tag SDS-PAGEである1, 2)。リン酸基補足分子であるアクリルアミド化Phos-tagを分離ゲル作成の際に添加してアクリルアミドと共重合させたゲルを用いる。タンパク質試料の調製法や電気泳動の操作・試薬は、すでに確立されたSDS-PAGE法と全く同じである。リン酸化タンパク質は、ゲルに固定されたPhos-tagと可逆的な結合を繰り返しながら泳動するため、相当する非リン酸化タンパク質よりも移動が遅れ、シフトアップしたバンドとして検出される(図1)。
本電気泳動法では、あるタンパク質のリン酸化されているアミノ酸残基数が同じであっても、その部位が異なる場合には、移動度の異なるバンドとして検出される。すなわち、リン酸化状態の違いを分離検出できる。セリン、スレオニン、チロシンのリン酸化はもちろん、酸性では加水分解を受けやすいヒスチジンやアスパラギン酸のリン酸化タンパク質も解析できるので、Phos-tag SDS-PAGEはこれらのリン酸基を介するバクテリアの情報伝達機構の研究の進展に寄与している3)。
また、Phos-tag SDS-PAGEプレキャストゲル、SuperSep Phos-tagシリーズも発売されているので、気軽に本法を研究に取り入れることができる。再現性が高くシャープなバンドが得られるプレキャストゲルを好んで継続的に利用している研究者も多い。筆者らのグループでもSuperSep Phos-tagを用いた例を報告しており4)、Phos-tag SDS-PAGEデータ集 (www.phostag.hiroshima-u.ac.jp) に画像を公開しているので、ご覧いただきたい。
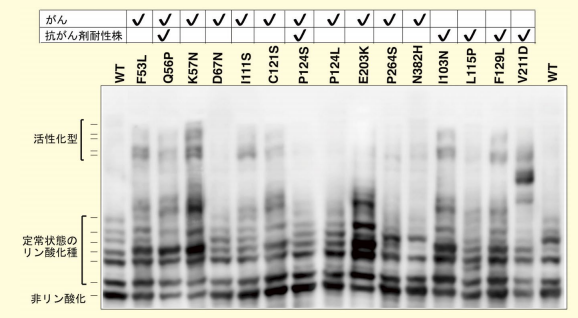
図2.がんにおけるMEK1変異とMEK1のリン酸化種の増加(文献5より転載)
肺がん、卵巣がん、および抗がん剤耐性細胞株で報告されている変異型MEK1の細胞内でリン酸化状態をPhos-tag SDS-PAGEで解析した。最近の筆者らのグループによるPhos-tag SDS-PAGEを用いた解析例として、がんにおけるMEK1の変異とMEK1のリン酸化亢進の関連を解析した研究を紹介する5)。肺がんや卵巣がんでは、細胞内情報伝達に関わるMEK1キナーゼの遺伝子に特徴的な変異が生じることが報告されている。MEK1遺伝子をクローニングし、報告された変異、あるいは抗がん剤耐性細胞株において報告された変異を導入したものを細胞に発現させ、そのリン酸化状態をPhos-tag SDS-PAGEを用いて解析すると、通常のMEK1では見られない、リン酸化種が存在することがわかる(図2)。
in vitroキナーゼアッセイで、これらの変異MEK1のキナーゼ活性が亢進していることも確認している。これらのことから、MEK1の変異が活性亢進の原因となり、がん化に繋がっていることが示唆された。
ビオチン化Phos-tagによるリン酸化タンパク質の解析法
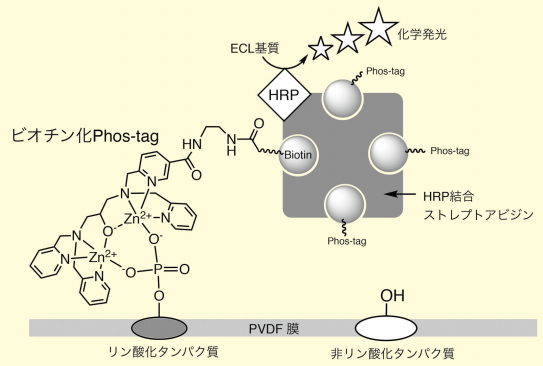
図3.ビオチン化Phos-tagを用いたPVDF膜上のリン酸化タンパク質検出の概略
2つ目は、PVDF膜上のリン酸化タンパク質を特異的に検出できるビオチン化Phos-tagである。ビオチン化Phos-tagを、予め市販品のHRP(西洋わさびペルオキシダーゼ)結合ストレプトアビジンと4:1の複合体を形成させておき、ブロット膜をプロービングする。ビオチン化Phos-tagと特異的に結合した膜上のリン酸化タンパク質は、市販の化学発光基質とHRPの反応(ECL)によって検出される(図3)。
これは一般的なウェスタン解析法に準じたものであり、使用するバッファー、試薬などは特別なものではなく、抗体をビオチン化Phos-tagに替えただけの簡便な方法である。
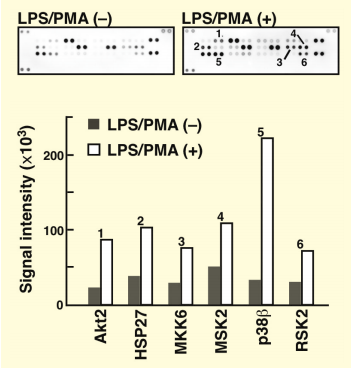
図4.LPS/PMAで刺激によってリン酸化するRaw264.7細胞内タンパク質の検出(文献6より転載)
抗体アレイ(Proteome Profiler Human Phospho-MAPK Array, R&D Systems) とビオチン化Phos-tagを用いてLPS/PMAで刺激したRaw264.7細胞抽出液中のリン酸化タンパク質を検出した。スポット1-6が陽性であり、刺激による有意なリン酸化が認められた。また、本法ではアレイ化されたペプチド基質のキナーゼアッセイ後のリン酸化検出、抗体アレイと反応したタンパク質のリン酸化解析など、アレイによる網羅的な検出にも利用できる。
図4に、Raw264.7細胞をLPS/PMAで刺激した際にAkt2、HSP27、MKK6、MSK2、p38β、RSK2などのリン酸化が亢進することを、抗体アレイとビオチン化Phos-tagを用いて検出した例を示す6)。
Phos-tagアガロースによるリン酸化タンパク質やリン酸化ペプチドの分離・濃縮
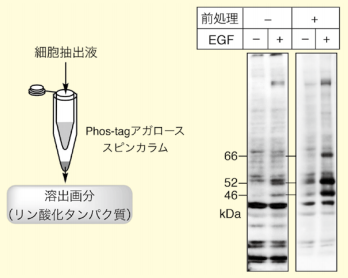
図5.Phos-tagアガローススピンカラムを用いたウェスタン解析サンプルの前処理によるリン酸化タンパク質の濃縮(文献8より転載)
A431細胞をEGF刺激し、SHCのリン酸化を抗リン酸化SHC抗体によるウェスタン解析した。Phos-tagアガロースカラムで前処理することによって、目的バンド(66, 52, 49 kDa)が鮮明に検出されるようになった。3つ目は、リン酸基親和性クロマトグラフィ担体、Phos-tagアガロースである。生体内に存在する全てのタンパク質のうち、60 %以上がリン酸化されうるが、それらは、ダイナミックに変化するキナーゼ/フォスファターゼ反応によって刻一刻とリン酸化と脱リン酸化を繰り返しており、実際にリン酸化状態にあるのは極微量である可能性が高い。そのようなリン酸化タンパク質を研究する上で、リン酸化タンパク質を分離、濃縮することは非常に有用である7)。
図5では、細胞抽出液をPhos-tagアガローススピンカラムで前処理することによって、抗リン酸化抗体によるウェスタン解析の特異的シグナルを増強させることができた例を紹介する8)。EGF刺激によるA431細胞内のSHCのリン酸化を抗リン酸化SHC抗体によってウェスタン解析する際に、リン酸化レベルが低い、あるいは抗体の特異性が高くないなどの原因で特異的シグナルが検出できなかったが、Phos-tagアガロースカラムによる前処理を行い、特異的シグナルが鮮明に検出できるようになった。
また、Phos-tagアガロースは質量分析を用いたリン酸化プロテオミクス解析における、リン酸化ペプチドの分離・濃縮にも有用である9)。
おわりに
Phos-tagを利用したことが記載されている研究論文件数は、2018年の1年間だけでも500件近くにのぼり(Google Sholarによる)、リン酸化解析のスタンダードとして広く浸透してきている。生命科学のトレンドの最先端分野でも利用されているのはもちろんだが、バクテリアや植物などの研究報告の比較的少ないタンパク質の解析に本法が利用されていることは特筆するべきことである。また、抗体が市販されていないタンパク質や、データベースを参照してもリン酸化の報告例がないマイナーなタンパク質の解析にも本法は適用できる。Phos-tag技術を広い分野の多くの研究者に利用していただき、Phos-tag製品がタンパク質のリン酸化解析に貢献できれば幸いである。
参考文献
- Kinoshita, E. et al. : Mol. Cell. Proteomics, 5, 749 (2006). DOI: 10.1074/mcp.T500024-MCP200
- Kinoshita, E. and Kinoshita-Kikuta, E. : Proteomics, 11, 319 (2011). DOI: 10.1002/pmic.201000472
- Kinoshita-Kikuta, E. et al. : PLoS One, 10, e0132598 (2015). DOI: 10.1371/journal.pone.0132598
- Kinoshita-Kikuta, E. et al. : Int. J. Chem., 4, e1 (2012). DOI: 10.5539/ijc.v4n5p1
- Kinoshita-Kikuta, E. et al. : Biochim. Biophys. Acta Proteins Proteom., 1867, 62 (2019). DOI: 10.1016/j.bbapap.2018.05.004
- Kinoshita, E. et al. : Anal. Biochem., 438, 104 (2013). DOI: 10.1016/j.ab.2013.03.029
- Kinoshita-Kikuta, E. et al. : Proteomics, 6, 5088 (2006). DOI: 10.1002/pmic.200600252
- Kinoshita-Kikuta, E. et al. : Anal. Biochem., 389, 83 (2009). DOI: 10.1016/j.ab.2009.03.026
- Yuan, E. T. et al. : Electrophoresis, 38, 2447 (2017). DOI: 10.1002/elps.201700175





