【総説】ncRNA の機能解析 ~結合タンパク質を求めて~
本記事は、和光純薬時報 Vol.84 No.3(2016年7月号)において、産業技術総合研究所 創薬分子プロファイリング研究センター 機能プロテオミクスチーム 足達 俊吾 様に執筆いただいたものです。

細胞にはリポソームRNAやトランスファーRNA、snoRNAなど、タンパク質をコードしないnon-coding RNA (ncRNA) が存在することは古くから知られていたが、次世代シークエンス技術に代表される近年の解析技術の発展により、以前に考えられていたよりもはるかに多くのncRNAが存在することが明かとなってきた1,2)。
また、その機能についても癌を含めた疾患との関係や、クロマチン状態制御、細胞内の構造体維持など多種多様な役割が明らかとなり、現在最も盛んに研究が行なわれている領域の一つとなっている3,4,5)。ncRNAの機能を解析するには、ncRNAのノックダウンやノックアウト、強制発現による細胞や個体への影響を確認する実験とともに、ncRNAが働く分子メカニズムを明らかとする必要がある(図1)。
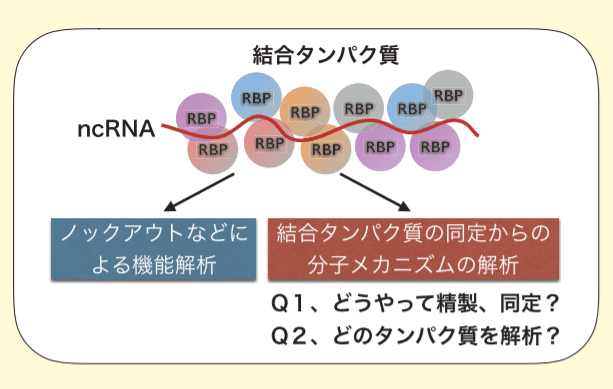
図1. ncRNA の解析の方向性と問題点
多くのncRNAは、タンパク質と結合することにより機能を発揮していると考えられていることから、ncRNAの機能を分子的に解明するためにはncRNAに結合するタンパク質を知ることがとても重要である。ncRNAに結合するタンパク質を知るためには、目的のncRNAとともにncRNA結合タンパク質を精製、質量分析を用いて同定する必要があるが、ここには大きな二つの問題がある。
一つ目は、目的のncRNA及びその結合タンパク質を精製する良い方法が確立されていないこと、二つ目は、ncRNAに限らず細胞内の長鎖のRNAには数百種類ものタンパク質が同時に結合しており、数百種類ものタンパク質を同時に同定する必要があることに加え、その中から実際に解析を行うタンパク質を選び出す必要があるが、その基準がないことである。
以下、これら二つの問題に対する我々のアプローチを紹介する(図1)。
RNA結合タンパク質の同定方法
我々はまず、特定のncRNAに結合するタンパク質を体系的に同定する方法の確立を目指した。ポジティブコントロールとして5種類のタンパク質(CDK9, CyclinT, Hexim, LARP7, MEPCE)の結合が知られている、7SK-RNAというncRNAを用い、結合タンパク質同定の確認実験を行った。
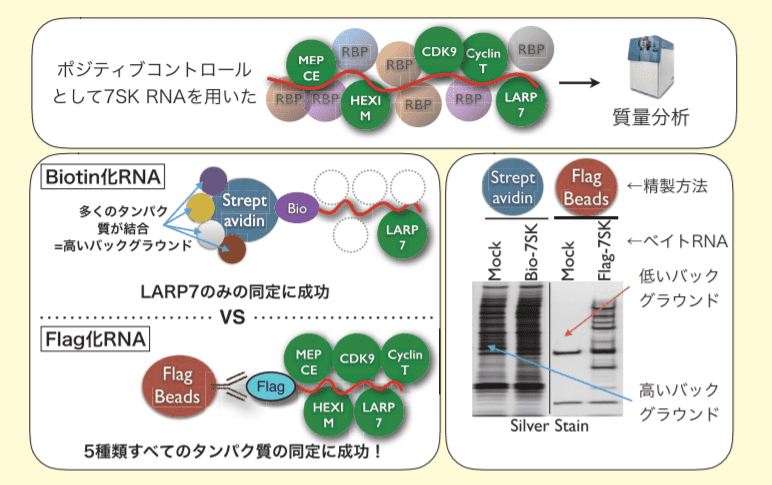
図2.7SK-RNA を例とした ncRNA 結合タンパク質の同定例
一般にRNAとタンパク質複合体の生成にはBiotin化RNA及び、ストレプトアビジン付加ビーズを用いた方法が知られていることから(図2)、我々は、Biotin標識化された7SK-RNAを in vitro 転写により合成、293T細胞ライゼートと混ぜ合わせ、ストレプトアビジン付加ビーズ(以下ストレプトアビジンビーズ)を用い7SK-RNA結合タンパク質を精製、質量分析を用い結合タンパク質を同定した。
その結果、残念なことにBiotin化RNA及び、ストレプトアビジンビーズを用いた精製方法では、LARP7以外の、CDK9, CyclinT, Hexim, MEPCEは同定されなかった。ストレプトアビジンビーズは非常に多くの細胞由来タンパク質と結合し、それらが精製のバックグラウンドとして存在すること(図2)、また質量分析には、夾雑タンパク質が多いと目的のタンパク質が夾雑タンパク質に埋もれ同定が困難になるという性質がある。
これらのことから我々は、既知7SK-RNA結合タンパク質の多くが同定できなかった原因としてストレプトアビジンビーズのバックグラウンドの高さにあると考え、新たなRNA及び結合タンパク質精製方法を模索、バックグラウンドが低いことが知られているFlag-peptide-tagと抗Flag抗体を用いた免疫沈降システム(以下Flagシステム)に注目した。
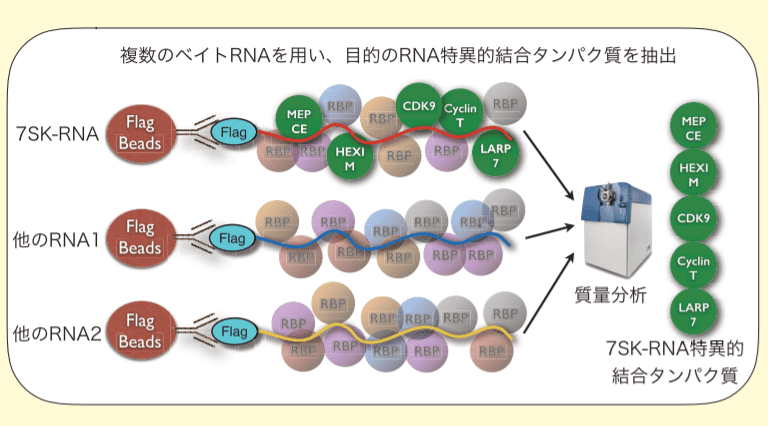
図3.比較による重要な RNA 結合タンパク質の同定
我々はFlagシステムをRNA結合タンパク質同定に応用するために、in vitro で合成したRNAの3'末端にFlagペプチドを化学的に付加(RNA-Flag)、抗Flag抗体を用いRNAを精製する方法を開発した(図2, 3)6)。
次に、7SK-RNAを含む複数のRNAについてRNA-Flag(ベイトRNA)を合成、ベイトRNAと293T細胞ライセートを混ぜ合わせ、抗Flag抗体を用いベイトRNA及び結合タンパク質を精製、質量分析を用いそれぞれのベイトRNAに結合するタンパク質を網羅的に同定した。
その結果、どのベイトRNAを用いた場合にも数百種類のタンパク質が同定されたが、既知の7SK-RNA結合タンパク質5種類については、7SK-RNAをベイトとして用いた場合のみに特異的に同定されていることが明らかとなった(図3)。
この結果は、目的のRNAを含む複数のベイトRNAを用意し、それぞれに結合するタンパク質を網羅的に同定、その中から目的RNA特異的に結合するタンパク質を抽出することにより、重要なRNA結合タンパク質が同定できる事を意味している。
我々はこの方法を用いLDLR-mRNAの3'UTR領域(広義のncRNA領域)に特異的に結合、LDLR-mRNAの安定性を制御するタンパク質としてZFP36L1/L2の同定にも成功6)、結合特異性を指標に重要なRNA結合タンパク質を同定するこの方法をiSRIM法(in vitro specificity based RNA regulatory protein identification method)と名付けた。
以上のように我々は、バックグラウンドの低いRNA精製方法の使用と、複数のベイトRNA間での結合タンパク質を比較することにより上記の2問題の解決に成功した。以下にそれぞれの実験上のポイントをまとめた。
バックグラウンドの少ないRNA精製のためのポイント
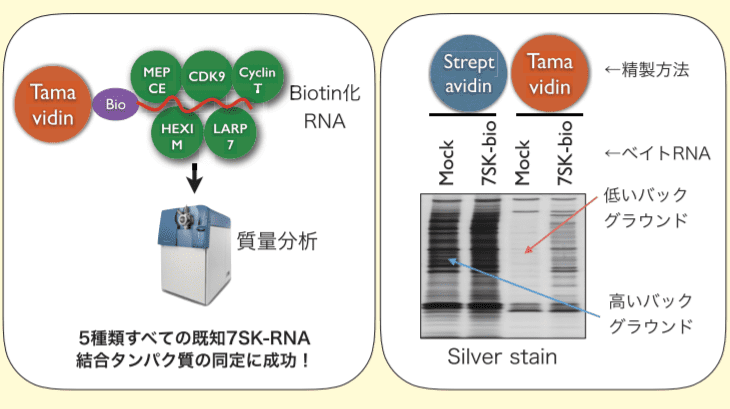
図4.Tamavidin magnetic beads を用いた解析例
バックグラウンドを少なくするための対策としては、上記のように精製システムそのものの選択も重要であるが、Biotin化RNAを用いる際でも別の方法が存在する。
我々は最近、和光純薬工業から販売されているTamavidin® 2-REV Magnetic Beads(以下Tamavidin®2)によるBiotin化分子の精製システムを用いた実験を行っている。Tamavidin®2はストレプトアビジンビーズに比べ、バックグラウンドが低く(図4)、実際にTamavidin®2を用いBiotin化7SK-RNAの結合タンパク質の同定実験を行ったところ、ストレプトアビジンビーズを用いた場合には同定が困難であった5種類の既知7SK-RNA結合タンパク質全ての同定に成功している。
Flag化RNAを用いる方法については、現状どの研究室でも使えるわけではないことから、RNA結合タンパク質の同定を考えておられる研究者にとって有用なツールであると考えられる。ただ、興味深いことにTamavidin®は、すべてのBiotin化分子と均一に結合するわけではなく、ある種のBiotin化分子について結合が弱くなることが知られており、我々の研究室でも確認している。
このことから、Tamavidin®2を用いる際にはまず、使用するBiotin化分子との結合を確認することをお勧めする。
また、特定の精製システムを用いるとしても精製に用いるビーズの種類によって大きく差が出るため、適切なビーズを選択する必要がある(例えば、一般的にはアガロースビーズよりも磁気ビーズのほうがバックグラウンドが少ない)。
そして、ビーズの量やベイト量の調節も重要な要素となりうる。ビーズ量が少なければ、バックグラウンドが減る一方、ビーズ量を減らしすぎると当然ながら精製できるベイトRNA量が減ってしまい、結合タンパク質の同定が困難となる。さらに、使用するベイト量が多すぎることもマイナスに働きうる。
これまでの経験上、RNA結合配列特異性の高いタンパク質(=目的のタンパク質)は結合配列特異性の低いタンパク質と比べ細胞内の存在量が少ない事が多く、一定以上のベイトRNA量を用いた場合、配列特異性の高いタンパク質の精製量は頭打ちとなり、配列特異性の低いタンパク質のみが増え、結果的に配列特異性の高いタンパク質(=目的のタンパク質)の同定を妨げることになる(筆者らは通常1×107個程度の細胞のライゼートを用いる際、細胞内のmRNAの総量よりはるかに少ない量である1pmol程度のベイトRNAを用いて実験を行っている)。
重要な結合タンパク質を選び出すためのポイント
上述のように、1種類のRNAには数百種類ものRNA結合タンパク質が結合していることから、同定された全てのタンパク質を解析することは不可能である。結合量の多いタンパク質から解析するという考え方もあるが、細胞内に多く存在し、RNA結合配列特異性の低いタンパク質は1種類のRNA上に複数結合している結果である可能性が高いため、ncRNAの機能としてRNAスポンジ7,8)などを想定している場合を除き、結合量の多いタンパク質を解析することは必ずしも賢明な策とも言えない。
我々は、上述のように複数のRNA間で結合タンパク質を比較し特異性を基準に解析すべきタンパク質を選ぶことが最も有効であると考えている。比較対象のRNAとしては例えば、目的ncRNAと明らかに機能が異なると考えられるncRNAや、目的ncRNAの相補配列、ncRNA上の機能エレメントが明らかな場合はその領域を欠損させた欠損変異体をベイトとして用いる事などが考えられるが、いずれの場合でも可能なかぎりベイトRNA量や長さが同じになるように配慮することが重要である。当然ながら、ビーズのみのmockとの比較は適切とは言えない。
今後の可能性
本稿では、ベイトRNAを合成し、in vitro においてベイトRNAと結合するRNA結合タンパク質を細胞ライゼート中から精製、同定する方法を紹介したが、最近、複数のアンチセンスオリゴを用いて内在性のRNAと結合するタンパク質を精製する方法として、CHART法やChIRP法などが開発され、実際にRNA結合タンパク質の同定に威力を発揮している9,10,11,12,13)。
このような実験においても、上述したポイントの重要性は変わらないか、特に目的ncRNAの長さが長い場合やncRNAの発現量が少ない場合は、より重要性が増すと考えられる。
キーワード
質量分析
分子の正確な質量を測定することにより、タンパク質を同定する方法。現在では一度の解析により数千タンパク質の同定、さらに定量が可能となっている。
Tamavidin®
タモギ茸より抽出されたBiotin結合タンパク質。Streptavidinと同様Biotin化分子の精製に用いられる。
7SK-RNA
細胞内に多量に存在する有名なncRNAの一つ。転写の活性に関わる。
参考文献
- Hirose, T. et al. : EMBO Rep., 15, 489 (2014). DOI: 10.1002/embr.201338390
- Cech, T. R. and Steitz, J. A. : Cell, 157, 77 (2014). DOI: 10.1016/j.cell.2014.03.008
- Ulitsky, I. et al. : Cell, 147, 1537 (2011). DOI: 10.1016/j.cell.2011.11.055
- Lee, J. T. : Science, 338, 1435 (2012). DOI: 10.1126/science.1231776
- Batista, P. J. and Chang, H. Y. : Cell, 152, 1298 (2013). DOI: 10.1016/j.cell.2013.02.012
- Adachi, S. et al. : Nucleic Acids Res., 42, 10037 (2014). DOI: 10.1093/nar/gku652
- Dickson, A. M. and Wilusz, C. J. : Wiley Interdiscip Rev RNA., 1, 173 (2010). DOI: 10.1002/wrna.18
- Tay, Y. et al. : Nature, 505, 344 (2014). DOI: 10.1038/nature12986
- Ideue, T. et al. : Proc. Natl. Acad. Sci. USA, 109, 5693 (2012). DOI: 10.1073/pnas.1200523109
- Simon, M. D. et al. : Nature, 504, 465 (2013). DOI: 10.1038/nature12719
- Quinn, J. J. et al. : Nat. Biotechnol., 32, 933 (2014). DOI: 10.1038/nbt.2943
- Engreitz, J. M. et al. : Cell, 159, 188 (2014). DOI: 10.1016/j.cell.2014.08.018
- Chu, C. et al. : Cell, 161, 404 (2015). DOI: 10.1016/j.cell.2015.03.025




