【総説】結晶構造から読み解くPAタグシステムの原理とタンパク質ループ構造への挿入
本記事は、和光純薬時報 Vol.85 No.1(2017年1月号)において、大阪大学 蛋白質研究所 高木 淳一先生、有森 貴夫先生、松永 幸子先生に執筆いただいたものです。

タンパク質の構造解析や機能解析を行うためには高純度の精製タンパク質が必要であり、筆者らの研究室ではより効率的な試料調製を行なうためのカスタムアフィニティータグシステムを開発してきた1,2)。中でもPAタグシステムは、12残基からなるPAタグ(GVAMPGAEDDVV)が、それに対するモノクローナル抗体NZ-1に対して極めて高い親和性で特異的に結合し(KD = 4 × 10-10 M)、しかもPAペプチドにより容易に競合溶出させることが可能なため、一段階で目的タンパク質を高純度かつ高収率で精製することができる3,4)。
そもそもPAタグシステムの開発は、東北大・加藤幸成教授らにより、悪性癌細胞に高発現する膜タンパク質ポドプラニンの部分配列(PAタグ配列)に対する特異的抗体NZ-1を取得したことに端を発する5)。NZ-1が単独のPAペプチドに対しても強く結合することに着目し、筆者らのグループによりアフィニティータグシステムとして確立するに至った。詳細は和光純薬時報Vol.82, No.4 (2014)および文献4を参照されたい。
最近、筆者らはPAペプチドとNZ-1の複合体の結晶構造を決定し、その特徴的な複合体形成機構より、PAタグシステムの新たな利用法を見出した6)。本稿ではPAタグのループ構造への挿入およびそのタンパク質機能解析ツールとしての応用例を報告する。
PAタグペプチド-NZ-1抗体の複合体構造解析
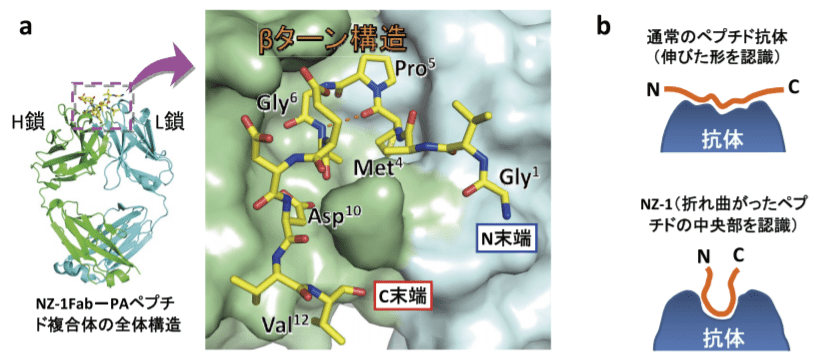
図1.NZ-1 による PA ペプチド認識機構
(a)NZ-1 - PA ペプチド複合体の全体構造と抗原結合部の拡大図。PA ペプチド(黄色のスティックモデル)は U 字型の構造をとり、その N 末端と C 末端の距離は近い。(b)抗ペプチド抗体によるペプチド認識モード。冒頭でも触れたが、PAタグはNZ-1へ高親和性で結合する。そのメカニズムを知るため、NZ-1のFabフラグメント単独およびPAペプチドとの複合体の結晶構造を決定した(PDB ID: 4YNY, 4YO0)。図1aに示すようにPAペプチドはMet4-Asp10を中心に分子全体でNZ-1抗体と接し、抗原認識部位の結合ポケットにすっぽりとはまり込んでいた。
また、Pro5-Gly6配列の部分ではβターン構造をとっていた。Pro-Gly配列は溶液中でもtypeⅡのβターン構造を形成しやすいことが知られていることから7)、PAペプチドはNZ-1と結合する前から結合に最適な構造を取ることができると予想される。抗体側の特徴もそれと類似し、ペプチドが結合する前後で立体構造および水和状態の変化がほとんど見られなかった。
すなわち、PAタグシステムにおいては、ペプチド側も抗体側もともに結合に伴うエントロピーの損失が非常に少ないことが結晶構造から示唆された。相互作用の親和性は結合前後のエネルギー差で決まるため、このようなエントロピー損失の少なさが、PAタグシステムが高い親和性を持つ主な要因であると考えられた。
また、NZ-1がU字型に曲がった構造のPAタグを認識することは、非常に興味深い知見であった。通常、ペプチドは溶液中で特定の構造をとらず、抗体に結合する場合も比較的伸びたコンフォメーションをとっていることが多い(図1b)。つまり、フラフラした状態でなければ抗体に認識されにくいため、既存のペプチドタグは、タンパク質のフレキシブルな領域、すなわちN末端あるいはC末端にしか付加できない場合が多い。
一方、PAタグは、NZ-1がPAペプチドの折れ曲がり構造を認識することから、PAペプチドのN末端とC末端は抗体に認識された状態では同じ方向を向き、距離も近くなる(図1b)。この特徴から、PAタグはタンパク質の末端だけでなく、Uターンしているループ部分へ挿入してもタグとして機能する、つまりNZ-1に認識されることが期待された。
PAタグのループ構造への挿入
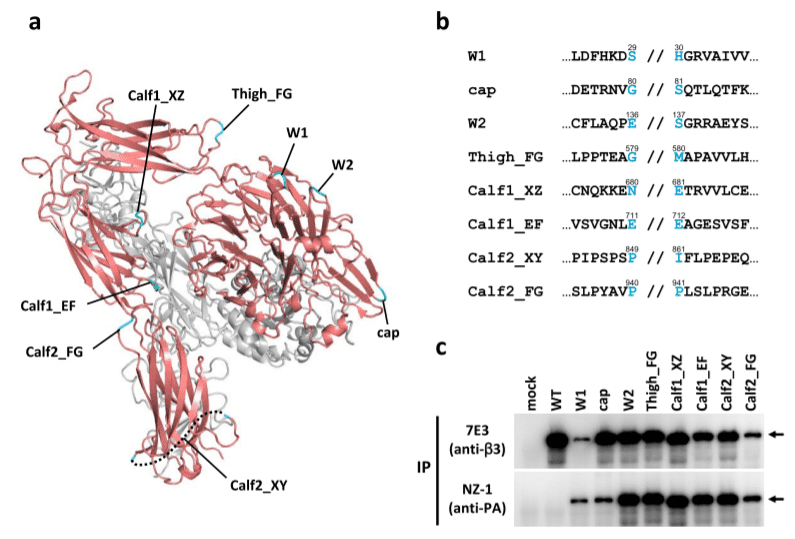
図2.インテグリンのループ領域への PA タグの挿入
(a)インテグリンαIIb(赤)β 3(灰色)の細胞外領域の立体構造(PDB ID: 3FCS)。PA タグを挿入したループ領域をシアンで示す。(b)挿入部位付近のアミノ酸配列。12 残基の PA タグは // の位置に挿入した。(c)各 PA タグ挿入変異体の、抗インテグリン抗体(7E3、上段)および抗 PA タグ抗体(NZ-1、下段)による免疫沈降の結果。文献 6 より改変して転載。PAタグ配列をループ構造へ挿入するためのテストサンプルとして、血小板膜タンパク質であるインテグリンを選んだ。インテグリンはα鎖とβ鎖のヘテロダイマーであり、αⅡb鎖とβ3鎖から構成されるインテグリンαⅡbβ3は立体構造や機能解析の報告例が多く、しかも細胞外領域のみでも可溶性タンパク質として高発現することから、PAタグ挿入の効果を評価するのに適した分子である8)。
まず、αⅡb鎖の分子表面に露出したループ構造(特にターン構造)を8ヵ所選び、PA配列12残基を挿入した変異体を作製した(図2a, 2b)。次に、これらPAタグ挿入αⅡb鎖変異体をβ3鎖と共に可溶性タンパク質として発現させたところ、NZ-1抗体および抗β3鎖抗体により免疫沈降されたことから、挿入PAタグはアフィニティータグとしての機能を保持しつつ、かつ、β3鎖とのヘテロダイマー形成に悪影響を与えないことが示された(図2c)。
同様の実験をFLAGタグやMycタグでも行ったが、これらでは(それぞれ抗FLAGタグのM2抗体及び抗Mycタグの9E10抗体に対して)まったく認識されなかったことから、ループに挿入できる能力はPAタグに特有のものであると言える6)。
この挿入できる性質がどのような場合に役立つのかを考えてみると、実にさまざまな用途が浮かんでくる。ひとつは、単純に、ターゲット分子の末端にタグを付加できない事情がある場合である。
N末端あるいはC末端残基が、まさに機能部位であったり活性部位と隣接していたりして、タグ付加を許さないタンパク質がいくつか知られている。そのようなタンパク質の一例として、神経軸索ガイダンス因子セマフォリン3Aが挙げられる。セマフォリン3Aは両末端が活性に寄与し、また複数の受容体との結合部位を持つため、「機能に影響のないループ」を選んで挿入するという目的に適した分子である。
実際、筆者らはセマフォリン3AのループにPAタグを挿入することで、高活性を持った試料の調製に成功している6)。このほかにも我々はこれまでに20種以上のタンパク質へのループ挿入を検討し、うまくデザインできればPAタグは比較的自由にループへ組み込めることがわかってきた。
挿入PAタグの分子レポーターとしての利用
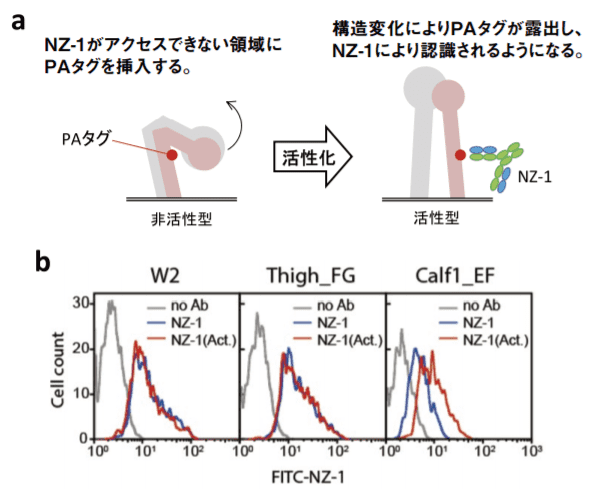
図3.インテグリンをターゲット分子とした PA タグループ挿入法の活用例
(a)PA タグシステムを用いてインテグリンの構造変化をモニター。(b)3 種のインテグリンα IIb 鎖 PA タグ挿入変異体の非活性状態(青)と活性状態(赤)のフローサイトメトリーヒストグラムを示す。文献 6 より改変して転載。ループに挿入したPAタグは、精製用タグとしてのみならず、分子の機能解析にも利用できるのではないだろうか。はじめにターゲット分子としてインテグリンα2b鎖を選択したのには前述の理由以外にも大きな理由がある。
インテグリンは構造変化に伴い活性が制御される分子であり9)、非活性状態(お辞儀構造)と活性状態(直立構造)では構造が大きく変化する。そこで、この構造変化の前後で露出度が変化する部位にPAタグを挿入し、NZ-1の結合量の変化により構造変化をモニターできるのではと考えた(図3a)。
このために、図2で用いたPAタグ挿入αⅡb鎖変異体のうち3種(W2、Thigh_FG、Calf1_EF)を細胞膜上に発現させ、フローサイトメトリーにより解析した。これらのうち非活性状態でも分子の外側にPAタグが位置するW2およびThigh_FGでは、インテグリンの活性化状態に関わらず同等の効率でNZ-1の結合が確認できた(図3b、W2およびThigh_FG、青色と赤色のヒストグラム)。
それに対して、PAタグがお辞儀構造の内側に位置するCalf1_EFにおいては、未刺激時(図3b、Calf1_EF、青色ヒストグラム)では他と比較してNZ-1により認識されにくく、あまり分子表面に露出していないことが予想された。ところがインテグリンを活性化し起き上がらせると(図3b、Calf1_EF、赤色ヒストグラム)、NZ-1の反応性は上昇した(ヒストグラムのピークが右へシフトした)。
このように、たった12残基のPAペプチド挿入により構造変化の追跡が可能となったわけである。
おわりに
本稿では、PAタグが単なる精製用タグとしてだけでなく、分子解析ツールとして応用できる一例を示した。他にも、PAタグは使い方次第で様々な解析に有用であると予想する。たとえば結晶構造解析においては、膜タンパク質などの結晶化が困難な分子の場合、フレキシブルな領域に結合する特異抗体の断片(Fab、単鎖抗体など)と複合体を形成させることにより結晶化を促進させる方法が主流となっているが、通常は、個々の標的タンパク質に対する抗体を取得する必要があり、多くの時間と労力を費やすことになる。
しかし、PAタグシステムでは好きな場所にタグを挿入し、NZ-1断片と複合体を形成させることで同様の効果が得られる可能性がある。これらの応用は、「高親和性」かつ「ループに挿入可能」という他にはない特異な性質を持つPAタグシステムだからこそ出来ることである。このような特長を生かし、多くの研究分野でPAタグシステムが応用されることを期待したい。
キーワード
ループ構造
ポリペプチド鎖の芳香を変える折れ曲がり構造。
ターン構造
3~4アミノ酸残基から成るU字型の二次構造。2端残基間の距離間隔や主鎖の二面角によって細分類される(α,β,γ,δ,π)。βターンが最も一般的。
参考文献
- Nogi, T. et al. : Protein Sci., 17, 2120 (2008). DOI: 10.1110/ps.038299.108
- Tabata, S. et al. : J. Proteomics, 73, 1777 (2010). DOI: 10.1016/j.jprot.2010.05.012
- Fujii, Y. et al. : Protein Expr. Purif., 95, 240 (2014). DOI: 10.1016/j.pep.2014.01.009
- 藤井勇樹 他:タンパク質科学会アーカイブ, 7, e075 (2014).
- Kato, Y. et al. : Biochem. Biophys. Res. Commun., 349, 1301 (2006). DOI: 10.1016/j.bbrc.2006.08.171
- Fujii, Y. et al. : J. Cell Sci., 129, 1512 (2016). DOI: 10.1242/jcs.176685
- Guruprsad, K. and Rajkumar, S. : J. Biosci., 25, 143 (2000).
- Zhu, J. et al. : Mol. Cell, 32, 849 (2008). DOI: 10.1016/j.molcel.2008.11.018
- Takagi, J. et al. : Cell, 110, 599 (2002). DOI: 10.1016/s0092-8674(02)00935-2




