Universal Sequencing Technology社 TELL-Seq™ Library Preparation Kit
TELL-Seq™は従来の短いDNAライブラリー解析(Short-read Sequencing)では解析が困難であったハプロタイプ解析やゲノム構造の解析が可能になります。
近年、長いDNAライブラリー解析(Long-read Sequencing)がShort-read Sequencingでは困難であった解析を行えることから注目されています。しかし、必要なサンプルの量が多く、また解析費用が高価であることやシーケンサーのトップシェアであるイルミナ機種で解析ができないという特徴があります。
TELL-Seq™は『Linked-read Sequencing』と呼ばれる疑似的に長いDNAライブラリーを形成することでLong-read Sequencingと同等の解析をイルミナ機種で行うことができます。(メーカーホームページはこちら)
Linked-Readとは
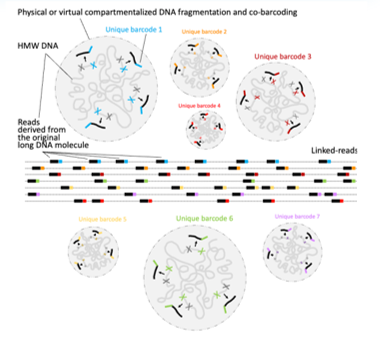
Linked-Readとは長いDNA配列を細分化し、それぞれの配列にユニークな認識バーコードがタグ付けし、それぞれの短い配列を決定した後に同じユニークバーコードの配列を「連結」することで長鎖DNAを再構築する解析方法です。
特長
- 安価 (Long-read Sequencingと比較)
- 操作時間が短い(3時間)
- 必要サンプル量が少ない(0.1 ~ 5ng)
ライブラリーの種類と特長
各ライブラリーのイメージ図
各ライブラリーの特長
| Short-read Seqencing | Long-read Sequencing | Linked-read Sequencing(TELL-Seq™) | |||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|
| 特長 | ・安価 ・イルミナ機種で解析可能 ・処理時間が短い ・解析の精度が高い |
・ゲノム構造を保持している ・解析の幅が広い |
・安価(Long-readと比較) ・イルミナ機種で解析可能 ・処理時間が短い ・解析の精度が高い ・ゲノム構造を保持している ・解析の幅が広い |
||||||||
| 問題点 | ・ゲノム構造を保持していない ・解析の幅が狭い |
・高価・専用の機種が必要 ・処理時間が長い・エラーが生じやすい |
・RNA-seqやCHIP-seqに対応していない | ||||||||
原理
TELL-Seq™はトランスポザーゼを用いた酵素法でゲノムDNAを断片化し、PCRによってアダプターを断片の両端に付与します。
-
トランスポザーゼによるDNA断片化 -
PCR後のライブラリー構造
解析
ソフトウェアのダウンロードは以下URLよりダウンロードできます。
https://universalsequencing.com/pages/tell-seq-software
解析ソフトは3種あります。
TELL-READ・・・シーケンサーのデータをfastqデータで抽出します。
TELL-SORT・・・マッピング解析での変異検出や、SV(Structure Variants)、フェーズ解析に使用します。
TELL-LINK・・・de novo assembly解析で使用します。
微小生物の解析例
微小生物のde novo assembly解析のデータ
様々な微小生物のゲノムをde novo assemblyで解析した結果。GC含有量が異なる生物間においてもゲノムの99%以上を解析できた(Genome fraction)。また、fastqから得られたデータでは長鎖DNA(High Molecule Weight DNA : HMW DNA)であることがわかった (Mean SLF size, NG50)。
Long-read Sequencingとの比較
大腸菌のゲノムをde novo assemblyで解析した結果。TELL-Seqは他社品と比べて必要なサンプル量が少量で済んでおり、Long-readと同等のコンティグであった。また、解析のエラーも低く、一塩基の欠失・挿入の割合も低かった(indels per 100kbp)。
ヒトゲノムの解析例
NA12878とNA24385由来のヒトゲノム解析
(A)NA12878とNA24385をそれぞれ2X146ペアエンド、もしくは2X96ペアエンドのシークエンスで解析した 結果。マッピング解析に十分なカバレッジ深度と長いコンティグが確認された。またエラーの割合も低い結果となった。
(B)TELL-Seq™によって調製をしたNA12878とNA24385のライブラリーをNoveSeqによって解析をした結果。NA12878とNA24385の両方でマッピングの整合性が確認できた。また、同じバーコードのライブラリーが繋がることで20kbより長いDNA断片が90%以上を占め、100kbの断片も20~30%確認された。
-
-
NA12878変異の検出感度
TELL-Seq™によって調製をした変異型NA12878の変異型ライブラリーをNoveSeqによって解析した結果。
一塩基の相違や一塩基の欠失や挿入などの一塩基の変異を高感度に検出できた。
-
-
de novoアッセンブリ解析
TELL-Seq™によって調製されたライブラリーをde novoアッセンブリ解析することで17番染色体のBRCA1の変異検出した。ハプロタイプ2で約1.5kbが欠失しているヘテロ接合体が確認された。
必要なキット
検体のゲノムサイズや数によって必要なキット数が変わってきます。
- 【Box】
- ①TELL-Seq WGS Library Reagent Box1 V1(#100035)
1Mb(base) – 200Mb 12サンプル/1個
200Mb – 1Gb 6サンプル/1個
1Gb – 5Gb 4サンプル/1個 - ②TELL-Seq WGS Library Reagent Box2 V1(#100036)
①と同じ - ③TELL-Seq™ Microbial Library Reagent Box 1, V1 (#100047)
1Mb – 50Mb 24サンプル/1個 - ④TELL-Seq™ Microbial Library Reagent Box 2, V1 (#100048)
③と同じ
- 【マルチプレックスプライマー】
- ⑤TELL-Seq Library Multiplex Primer(#553-41381)
TELL-Seq Library Multiplex Primer(#100009)
TELL-Seq Library Multiplex Primer(#100010)
TELL-Seq™ Library Multiplex Primer C-series (1-96) Plate*(#100043)
*多検体での解析を行う方にオススメです。
- 【イルミナシーケンサープライマー】
- ⑥TELL-Seq Illumina Sequencing Primer(#550-41391)
必要キットの例
例1)200Mbの細菌ゲノムを同時に8サンプル解析したい
【必要キット】
①×1, ②×1, ⑤×1種類, ⑥×1
例2)3Gbのヒト全ゲノムを同時に9サンプル解析したい
【必要キット】
①×3, ②×3, ③×2種類, ④×1
例3)1Gbのゲノムを同時に18サンプル解析したい
【必要キット】
①×3, ②×3, ③×3種類, ④×1
例4)40Mbの細菌ゲノムを同時に10サンプル解析したい
【必要キット】
②×1, ③×1,⑤×2種類, ⑥×1
技術的なお問い合わせ
TELL-Seq™に関する技術的なご質問は製造元であるUST社のホームページからもお問い合わせができます。
早急に回答が欲しい際はご活用ください。
*ご質問の言語は英語となります。
*当社経由でのご質問も承っております。
製品一覧
- 項目をすべて開く
- 項目をすべて閉じる
TELL-Seq Box
TELL-Seq Microbial Box
TELL-Seq Multiplex Primer
TELL-Seq Illumina Sequencing Primer
- 掲載内容は本記事掲載時点の情報です。仕様変更などにより製品内容と実際のイメージが異なる場合があります。
- 掲載されている製品について
- 【試薬】
- 試験・研究の目的のみに使用されるものであり、「医薬品」、「食品」、「家庭用品」などとしては使用できません。
- 試験研究用以外にご使用された場合、いかなる保証も致しかねます。試験研究用以外の用途や原料にご使用希望の場合、弊社営業部門にお問合せください。
- 【医薬品原料】
- 製造専用医薬品及び医薬品添加物などを医薬品等の製造原料として製造業者向けに販売しています。製造専用医薬品(製品名に製造専用の表示があるもの)のご購入には、確認書が必要です。
- 表示している希望納入価格は「本体価格のみ」で消費税等は含まれておりません。
- 表示している希望納入価格は本記事掲載時点の価格です。



